轉(zhuǎn)座子整合穩(wěn)定細(xì)胞株
▏ 名字定義
轉(zhuǎn)座因子(Transposable element,簡(jiǎn)稱TE),又稱“跳躍基因”,是指從基因組的一個(gè)位置移動(dòng)到另一個(gè)位置的DNA序列。這類因子最早由遺傳學(xué)家芭芭拉·麥克林托克于50多年前發(fā)現(xiàn)。起初,生物學(xué)家對(duì)麥克林托克的發(fā)現(xiàn)持懷疑態(tài)度。然而,在接下來的幾十年里,人們逐漸發(fā)現(xiàn),TE不僅會(huì)“跳躍”,而且?guī)缀醮嬖谟谒猩矬w(包括原核生物和真核生物)中,而且通常數(shù)量巨大。例如,TE約占人類基因組的50%,占玉米基因組的90%。
▏ 轉(zhuǎn)座子類型
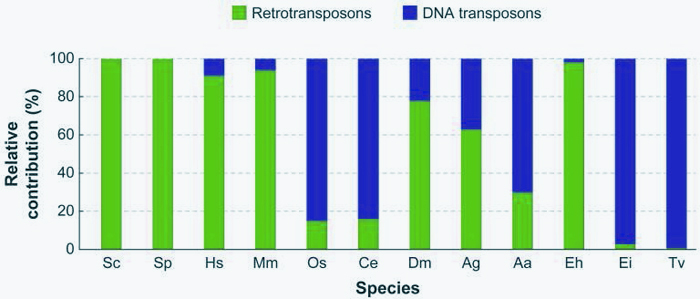
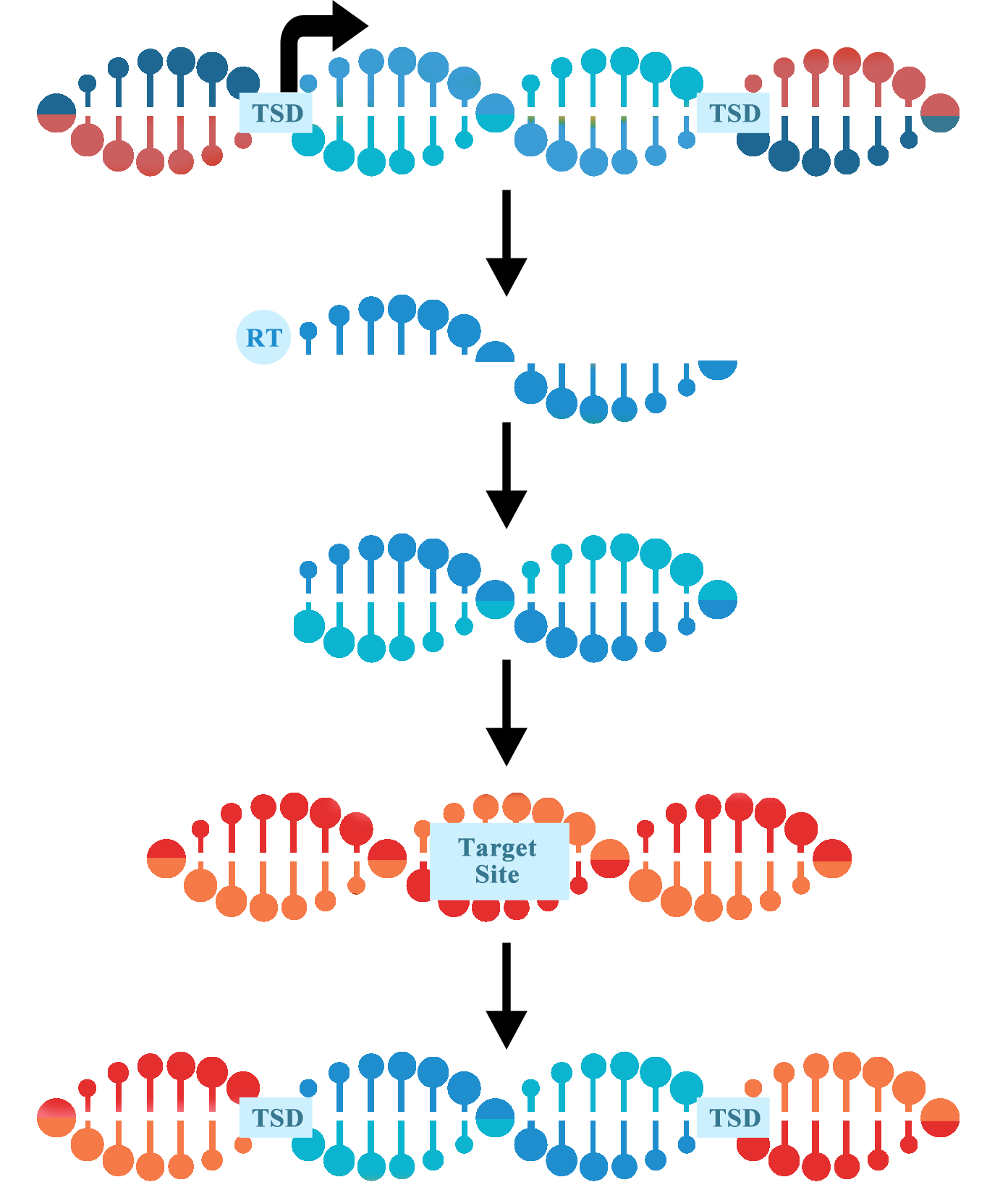
圖 1:逆轉(zhuǎn)錄轉(zhuǎn)座子轉(zhuǎn)座概述。逆轉(zhuǎn)錄轉(zhuǎn)座子通過“復(fù)制粘貼Ctrl C+V”機(jī)制進(jìn)行移動(dòng)。
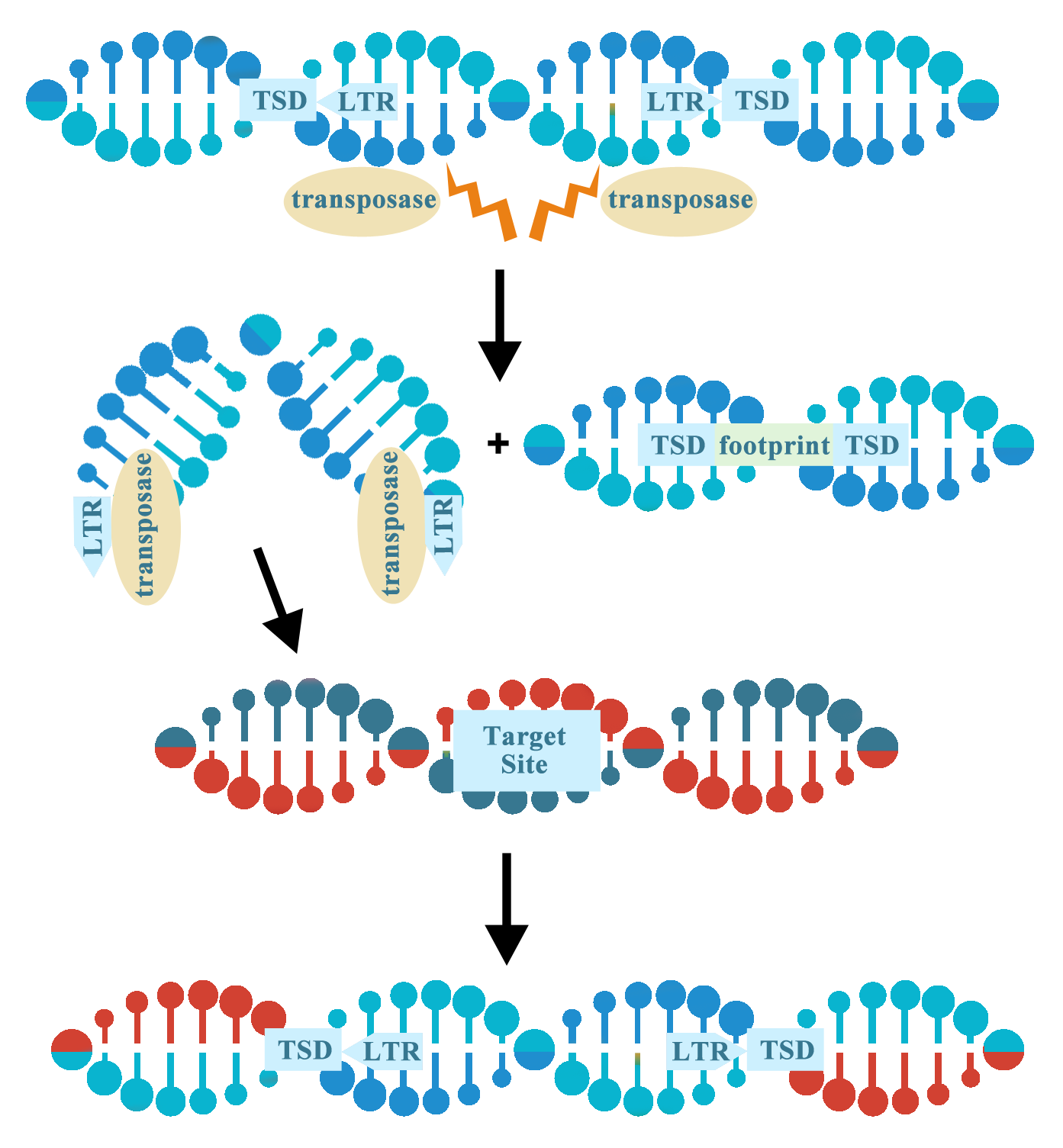
▏ 實(shí)驗(yàn)室中常用的DNA轉(zhuǎn)座子
雖然轉(zhuǎn)座子有很多不同的類型,但DNA轉(zhuǎn)座子在實(shí)驗(yàn)室中用于基因組操作最為常見。在實(shí)驗(yàn)室中使用轉(zhuǎn)座子時(shí),轉(zhuǎn)座酶基因以反式形式提供,以便將目標(biāo)基因插入轉(zhuǎn)座子的長(zhǎng)末端重復(fù)序列(LTR)之間,類似于病毒載體的包裝過程。
常見的三種適合用作研究工具的轉(zhuǎn)座子系統(tǒng):睡美人Sleeping beauty、PiggyBac和Tol2。
1.睡美人Sleeping beauty,簡(jiǎn)稱SB
睡美人是一種合成的轉(zhuǎn)座因子,由魚類中發(fā)現(xiàn)的mariner。睡美人首選的整合靶位是TA二核苷酸,被轉(zhuǎn)座酶切割后,它會(huì)在切除位點(diǎn)的末端序列上留下CAG DNA足跡。它的貨物容量>100 kB,但整合效率會(huì)隨著貨物大小而降低。睡美人通過哺乳動(dòng)物基因組進(jìn)行整合,其整合特征接近隨機(jī)。SB在脊椎動(dòng)物中很活躍,在人類細(xì)胞中的整合速度與逆轉(zhuǎn)錄病毒載體相似。SB轉(zhuǎn)座酶的高活性版本SB100X的效率比第一代SB轉(zhuǎn)座酶高~100 倍。 hySB100x在SB100X的基礎(chǔ)上進(jìn)行了改進(jìn),轉(zhuǎn)座活性提高了 30%。
2.piggyBac
piggyBac是在菜青蟲中發(fā)現(xiàn)的 ,它的靶位是 TTAA,與其他轉(zhuǎn)座子不同,它在切除后不會(huì)留下 DNA 足跡序列。piggyBac可以裝載超過100 kB的DNA,并且在酵母、植物、昆蟲和哺乳動(dòng)物細(xì)胞(包括人類細(xì)胞)中體外和體內(nèi)均有活性。piggyBac傾向于在轉(zhuǎn)錄起始位點(diǎn)、CpG 島和 DNaseI 高敏位點(diǎn)進(jìn)行整合。與睡美人一樣,piggyBac在人類細(xì)胞中的整合效率與逆轉(zhuǎn)錄病毒載體相似。與密碼子優(yōu)化的野生型piggyBac轉(zhuǎn)座酶相比,高活性PB轉(zhuǎn)座酶 (hyPB)在哺乳動(dòng)物細(xì)胞中的活性高約10倍。
3.Tol2
Tol2是第一個(gè)在脊椎動(dòng)物中報(bào)道的活性 DNA 轉(zhuǎn)座子。它是在日本青鳉魚中發(fā)現(xiàn)的,因?yàn)樗迦氲角圜汈~的酪氨酸酶基因中導(dǎo)致了白化病。與睡美人(Sleeping Beauty)和piggyBac不同,Tol2 的首選整合位點(diǎn) TNA(C/G)TTATAA(G/C)TNA 具有較弱的共識(shí)序列。Tol2 可以將10-11 kB的DNA 遞送到哺乳動(dòng)物細(xì)胞中而不會(huì)降低效率,最大載量約為200 kB。與piggyBac類似,Tol2也傾向于在轉(zhuǎn)錄起始位點(diǎn)、CpG 島和 DNaseI 高敏位點(diǎn)進(jìn)行整合。Tol2 僅在脊椎動(dòng)物中活躍,在人類細(xì)胞中的整合效率低于piggyBac和睡美人。Minimal Tol2或miniTol2是原始Tol2的截短版本,其轉(zhuǎn)座活性提高了約 3 倍。
|
|
Sleeping Beauty (SB) |
piggyBac (PB) |
Tol2 |
|
Species of origin |
Salmonid fish |
Cabbage looper moth |
Medaka fish |
|
Classification |
Tc1/mariner superfamily |
PB superfamily |
hAT superfamily |
|
Transposable element |
~1.6 kb long |
~2.5 kb long |
~4.7 kb long |
|
Terminal regions |
IR/DRs of ~ 230 bp |
35–63 bp with outer TIRs and inner subterminal IRs |
150–200 bp containing the TIRs and subterminal regions |
|
Transposase |
340 aa |
594 aa |
649 aa (most active isoform) |
|
Footprint |
CAG |
None |
Variable |
|
Target site preference |
TA |
TTAA |
Weak consensus sequence |
|
TNA(C/G)TTATAA(G/C)TNA |
|||
|
Target site duplication |
TA |
TTAA |
8 bp |
|
Activity in species |
Various vertebrates |
Vertebrates, insects, plants, yeast |
Various vertebrates |
|
Efficiency in human cells |
Comparable to retroviral vectors |
Comparable to retroviral vectors |
Lower than PB and SB |
|
Cargo capacity |
>100 kb |
>100 kb |
>100 kb |
|
Overproduction inhibition |
Yes |
To some extent |
Lower than PB and SB |
|
Integration profile |
Close-to-random |
Biased towards TSSs, CpG islands and DNaseI hypersensitivity sites |
Biased towards TSSs, CpG islands and DNaseI hypersensitivity sites |
|
Most common parental plasmid |
pT2 |
pXL-BacII |
pTol2, miniTol2 |
|
Most hyperactive transposase |
hySB100X |
hyPB |
hTol2-M |
|
Vectors for transposon delivery |
Plasmid DNA, pFAR, MC, |
Plasmid DNA, dbDNA, |
Plasmid DNA |
|
non-integrative viral vectors, nanoparticles |
non-integrative viral vectors, nanoparticles |
||
|
Vectors for transposase delivery |
Plasmid DNA, mRNA, SNIM RNA, |
Plasmid DNA, mRNA, |
Plasmid DNA, mRNA, |
|
recombinant protein (hsSB), non-integrative viral vectors, |
non-integrative viral vectors, |
recombinant protein (His-Tol2) |
|
|
nanoparticles |
nanoparticles |
|
|
|
Clinical trials |
Yes |
Yes |
No |
▏ 常見應(yīng)用
1.轉(zhuǎn)座子誘變篩選:
轉(zhuǎn)座子本質(zhì)上是一種致突變?cè)@使得它們成為檢測(cè)功能喪失或功能獲得突變的誘變篩選的絕佳工具。在這些篩選中,轉(zhuǎn)座子編碼報(bào)告基因、誘變盒或條形碼。當(dāng)轉(zhuǎn)座子被遞送到細(xì)胞或模型生物中時(shí),它們會(huì)插入宿主的基因組中。然后,使用下一代測(cè)序檢測(cè)轉(zhuǎn)座子插入位點(diǎn),并進(jìn)行分析以確定在實(shí)驗(yàn)過程中哪些插入是陽性或陰性選擇的。
2.轉(zhuǎn)基因動(dòng)物:
轉(zhuǎn)基因動(dòng)物通常是通過將DNA直接注射到受精卵的原核中產(chǎn)生的,這會(huì)導(dǎo)致該序列隨機(jī)地整合到受精卵的基因組中,這個(gè)過程高度難以預(yù)測(cè)。然而,轉(zhuǎn)座子在注射到受精卵的細(xì)胞質(zhì)中后,能夠有效地整合到受精卵的基因組中,而當(dāng)DNA直接注射時(shí),這個(gè)過程效率很低。睡美人、 piggyBac和Tol2都已被用于生成轉(zhuǎn)基因動(dòng)物,包括斑馬魚、小鼠、大鼠和兔子。
3.穩(wěn)定細(xì)胞株構(gòu)建
轉(zhuǎn)座子是慢病毒載體構(gòu)建穩(wěn)定細(xì)胞株的替代方案,例如用于iPSC 重編程以及基因和細(xì)胞治療,并且有可能克服病毒的一些局限性。TE 的有效載荷很大,使用Sleeping Beauty和piggyBac時(shí)可達(dá)100 kB,這比病毒載體有很大優(yōu)勢(shì)(AAV 的有效載荷約為 5 kB,慢病毒的有效載荷約為 8 kB)。轉(zhuǎn)座子也比病毒載體更不容易誘導(dǎo)免疫反應(yīng),并且也更容易和更便宜地生成。兩種傳遞方法都可能發(fā)生因整合而導(dǎo)致的基因破壞,但由于 TE 主要插入基因間區(qū)域,因此基因破壞不太令人擔(dān)憂。
4.SgRNA引導(dǎo)的定點(diǎn)轉(zhuǎn)座
可以借助于sgRNA來引導(dǎo)定點(diǎn)位置的轉(zhuǎn)座,有報(bào)道稱,INTEGRATE系統(tǒng)(引導(dǎo) RNA 輔助靶向插入轉(zhuǎn)座因子)可在細(xì)菌中實(shí)現(xiàn)高達(dá)10 kB大小的 DNA 的約 100% 整合。
▏ 穩(wěn)定細(xì)胞株構(gòu)建服務(wù)
科佰生物提供“轉(zhuǎn)座子整合系統(tǒng)”穩(wěn)定細(xì)胞株構(gòu)建服務(wù),利用該系統(tǒng),我們已經(jīng)成功構(gòu)建超過400株穩(wěn)定細(xì)胞株的模型,有著豐富的經(jīng)驗(yàn),歡迎溝通咨詢。
|
|
質(zhì)粒轉(zhuǎn)染 |
病毒感染 |
Flp-FRT系統(tǒng) |
轉(zhuǎn)座子系統(tǒng) |
基因編輯 |
||
|
脂質(zhì)體 |
電轉(zhuǎn) |
慢病毒 |
|||||
|
適用細(xì)胞 |
部分細(xì)胞 |
幾乎所有細(xì)胞 |
幾乎所有細(xì)胞 |
內(nèi)部4種細(xì)胞 |
幾乎所有細(xì)胞 |
部分細(xì)胞 |
|
|
轉(zhuǎn)染/感染效率 |
低 |
中 |
高 |
高 |
高 |
低 |
|
|
隨機(jī)整合 |
是 |
是 |
是 |
否 |
- |
否 |
|
|
生物安全等級(jí) |
BL1 |
BL1 |
BL2 |
BL1 |
BL1 |
BL1 |
|
|
基因裝載能力 |
- |
- |
<4-5k |
- |
- |
- |
|
|
陽性率 |
低 |
高 |
高 |
高 |
高 |
低 |
|
|
周期 |
基因合成 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
2-3周 |
|
病毒包裝 |
- |
- |
1周 |
- |
- |
- |
|
|
混合克隆篩選 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
1-2周 |
|
|
單克隆篩選 |
>3 周 |
>3 周 |
>3 周 |
可選 |
可選 |
>3 周 |
|
|
合計(jì) |
> 8-10 周 |
> 8-10 周 |
>9-11周 |
5-7 周 |
5-7 周 |
> 8-10 周 |
|
|
工作負(fù)荷 |
高 |
中 |
中 |
低 |
低 |
高 |
|


